El género Salmonella pertenece a la familia Enterobacteriaceae. Son bacilos gram negativos, de 0,7-1,5 x 2-5 μm, anaerobios facultativos, no formadores de esporas, generalmente móviles por flagelos perítricos. Las bacterias crecen óptimamente a 37°C y pueden catabolizar la D-glucosa y otros carbohidratos con producción de ácido y gas.
Fermentan glucosa, maltosa y manitol, pero no fermentan lactosa ni sacarosa. Son generalmente catalasa positiva, oxidasa negativa y reducen nitratos a nitritos. La mayoría de estas características se utilizan para la identificación bioquímica de cepas aisladas de Salmonella.
-
Objectivos
Analizar una muestra de agua contaminada (Residual Urbanas). Incubar Salmonella en dos etapas de pre-enriquecimiento, enriquecimiento y luego sembrar en agar XLD. Y ver si se forman colonias de Salmonella.
-
Fundamento
Primero haremos preenriquiment no selectivo de la muestra de agua con agua de peptona. Con ello conseguimos el crecimiento de aquellas colonias de Salmonella que puedan estar lesionadas, incrementando su vitalidad y adquiriendo las condiciones fisiológicas necesarias para su óptimo crecimiento y desarrollo posterior. Este medio no es inhibidor de la flora acompañante.
Después haremos el enriquecimiento selectivo en caldo Rappaport-Vassiliadis que es un medio diferencial. Contiene peptona como fuente de carbono y nitrógeno, MgCl2 para elevar la presión osmótica del medio, ver malaquita como inhibidor de otros microorganismos, el pH de medio se (5,1 ± 0,2). El pH bajo, combinado con la presencia de verde malaquita y la alta concentración de MgCl2, incrementa la presión osmótica, tiene carácter selectivo para Salmonella, ya que es capaz de crecer en estas condiciones respecto a otras enterobacterias, aunque otras enterobacterias pueden crecer ligeramente como Shigella.
Al final haremos una siembra en placa en agar XLD (Xilosa-Lisina-Desoxicolat), un medio selectivo y diferencial utilizado para la diferenciación de enterobacterias especialmente de Salmonella y Shigella. El agar XLD contiene extracto de levadura, lactosa y sacarosa como fuente de nutrientes y vitaminas; utiliza el desoxicolat de sodio como agente selectivo, inhibiendo los microorganismos gram positivos. La xilosa se incorpora en el medio dado que la fermentan prácticamente todos los enterobacterias, produciendo ácido y haciendo girar el rojo de fenol en color amarillo.
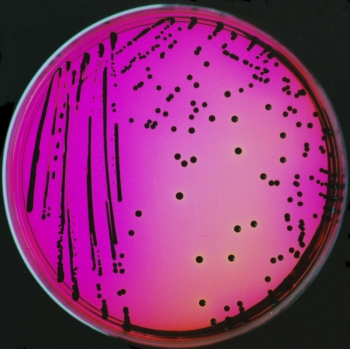
En este medio Los enterobacterias fermentan xilosa produciendo ácido y se quedan de color amarillo. La Shigella únicamente utiliza Lisina y se queda de color rojo. Salmonella primer fermenta xilosa quedando de color amarillo, cuando termina xilosa comienza a utilizar Lisina y se queda de color rojo. Por eso se pone tiosulfato de sodio en medio que salmonela puede utilizar produciendo H2S que reacciona con citrato férrico produciendo un precipitado negro. Así podemos distinguir las colonias de Salmonella de color negro.
Los colores finales son: (Enterobacterias = Amarillo / incolora) / (Shigella = Rojo) / (Salmonella = Negro (Figura 1))
-
Materiales
Tubos de ensayos, placas de petri, asa de siembra, mechero bunsen, micro-pipeta, pipum, pipeta graduada.
-
Reactivos
Agua de Peptona: El agua de peptona tamponada se utiliza para el pre-enriquecimiento no selectivo de Salmonella. Contiene: Peptona, Cloruro de sodio, Fosfato disódico y Fosfato mono potásico.
Solución Ringer: Solución isotónica que sirve para la conservación de la bacteria.
Caldo Rappaport-Vassiliadis: Es un medio líquido para el enriquecimiento selectivo de Salmonella a partir de carne
vacuna y productos lácteos, heces y agua contaminada. Contiene: Triptona, Cloruro sódico, Fosfato mono potásico, Cloruro de magnesio anhidro y Oxalato de verde malaquita.
Agar XLD: El Agar XLD (Agar Xilosa, Lisina y Desoxicolato) es un medio moderadamente selectivo y diferencial para el aislamiento y diferenciación de microorganismos Gramnegativos entéricos patógenos (Salmonella y Shigella).
-
Procedimiento
- Pre-enriquecimiento
I. Poner 20ml de agua de peptona 20g/L en tres tubos de ensayo-
II. Poner estos tubos en autoclave a 121ºC para 15 minutos para esterilizar.
III. Mientras tanto encender mechero bunsen para asegurar zona de esterilidad de unos 40cm de radio.
IV. Después de 15 minutos sacar los tubos de autoclave y dejar enfriar en zona de esterilidad.
V. Mientras los tubos de agua de peptona están enfriando hay que preparar otros 3 tubos en condiciones de esterilidad.
a) Poner 10ml de agua de problema y rotular “Muestra Directa” o (MD)
b) Poner 9ml de solución Ringer y 1ml de “Muestra Directa” y rotular (1/10).
c) Poner 9ml de solución Ringer y 1ml de tubo (1/10) y rotular (1/100).
VI. De tubo de (“MD”,”1/10″ y “1/100”) coger 1ml de inoculo con micropipeta y poner en tubos de agua de peptona y marcar con dilución correspondiente.
VII. Poner los tubos de agua de peptona en un vaso de precipitado y dejarlos en estufa a 37ºC para 18h.
- Enriquecimiento
I. Poner en 3 tubos de ensayo 10ml de caldo Rappaport-Vassiliadis en condiciones de esterilidad.
II. Sacar los tubos de agua de peptona de estufa.
III. Coger 1ml de tubos de agua de peptona y poner en tubos de caldo R-V y marcar la dilución correspondiente.
IV. Dejar los tubos de caldo R-V en estufa a 42ºC para 24h.
- Siembra en Agar XLD
I. Coger 3 placas de Petri y poner agar XLD en la placa Petri.
II. Dejar solidificar el agar en condiciones de esterilidad.
III. Cuando el agar esta frio coger un inoculo de tubos de caldo R-V con nansa de siembra y sembrar sobre las placas en forma de triple estaría.
IV. Dejar las placas en estufa a 37ºC para 24 horas.
– Resultados positivos de Salmonella en Agar XLD. (Figura 1). Los resultados se dan en agua positiva o negativa en Salmonella.